시작하며
안녕하세요, 한주현입니다.
오늘은 bed 파일을 ucsc genome browser에 추가하는 방법에 대해 알아보겠습니다.
| 목차 |
|
bed 파일 준비하기 genome browser에 넣기 |
bed 파일 준비하기
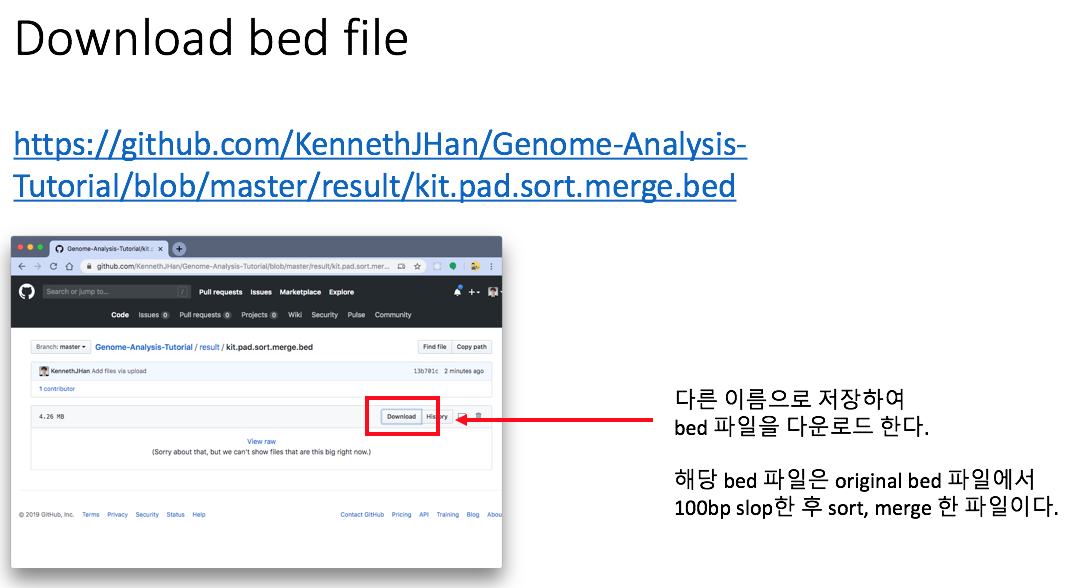
다음 예시 링크에 들어가 bed 파일을 다운받습니다.
https://github.com/KennethJHan/Genome-Analysis-Tutorial/blob/master/result/kit.pad.sort.merge.bed
genome browser에 넣기
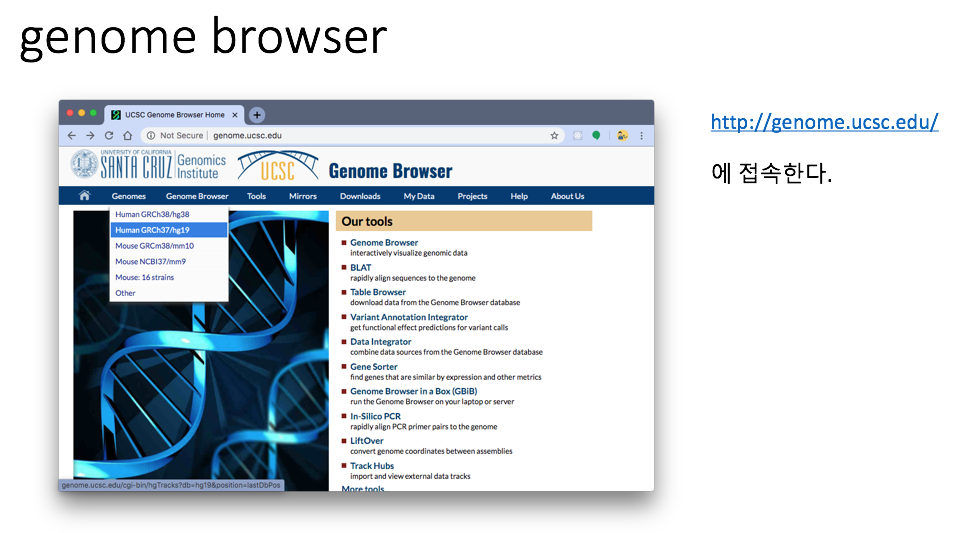
1) http://genome.ucsc.edu 에 접속하여 Genomes의 Human GRCh37/hg19 을 클릭한다.
2) 다음 화면의 아래에 add custom tracks 버튼을 클릭한다.

3) Choose File 버튼을 누른다. 창이 뜨면 bed 파일을 넣어주고 업로드를 기다린다.

4) 빨간 화살표가 가리키는 부분에 파일 이름이 나오면 파일이 제대로 업로드 된 것이다.
그 다음 Submit 버튼을 누른다.

5) Track의 이름을 바꿀 수 있는데 User Track 링크를 누르면 된다.
바꾸고 싶지 않다면 오른쪽의 go 버튼을 눌러도 된다.

6) 다음 그림의 빨간 네모 친 부분을 바꾸고 Submit 버튼을 누르면 된다.

7) 바뀐 모습

8) go 버튼을 눌러 genome browser로 이동한다.

9) custom track이 genome browser에 추가되었다. 다음 그림은 chr15:48426484 를 검색한 것이다.

10) 100x 를 두 번 눌러 zoom out 하여 bed가 genome 의 어느 부분에 구간을 잡고 있는지 확인할 수 있다.

오늘은 genome browser에 bed 파일을 추가하는 방법에 대해 알아보았습니다.
여러분들께 도움이 되셨음 좋겠습니다.
그럼 다음 포스트에서 만나요~
'생물정보학 > Tools' 카테고리의 다른 글
| [생물정보학] ORF와 CDS의 차이점과 refGene.txt.gz 파일 (0) | 2020.07.05 |
|---|---|
| [생물정보학 유튜브 강의] 생물정보학 툴 설치 (samtools) 및 bam 파일 보는 방법 (1) | 2020.06.14 |
| VCF GT, DP, AD와 genotype 0/0 , 0/1 , 1/1의 의미 (1) | 2019.07.15 |
| [github] github 파일 다운로드 방법 (5) | 2019.05.27 |
| [Tool] BLAST 리눅스 로컬 설치 및 실행방법 - BLAST LINUX local install and execute - BLAST 로컬 설치의 장단점 (2) | 2018.12.08 |



댓글