안녕하세요 한주현입니다.
오늘은 github에 있는 파일을 다운 받는 방법에 대해 알아보겠습니다.
들어가며
제가 출판한 "바이오파이썬으로 만나는 생물정보학" 도서 뿐만 아니라 요새 출판되는 여러 도서 및 소스코드 공유는 github로 많이 제공되고 있습니다.
간간히 저에게 자료를 어떻게 받는지 방법에 대해 문의하시는 분들이 계셔서
오늘은 github에서 자료를 받는 세 가지 방법에 대해 알아보겠습니다.
1. 그냥 사이트에 들어가서 하나씩 받기
다음 주소에 있는 파일인 blastSample.fasta 파일을 하나 받아보겠습니다.
https://github.com/KennethJHan/Bioinformatics_Biopython/blob/master/Section1/Chap1/blastSample.fasta
링크에 들어가시면 다음 사진과 같이 나오게 되는데요..
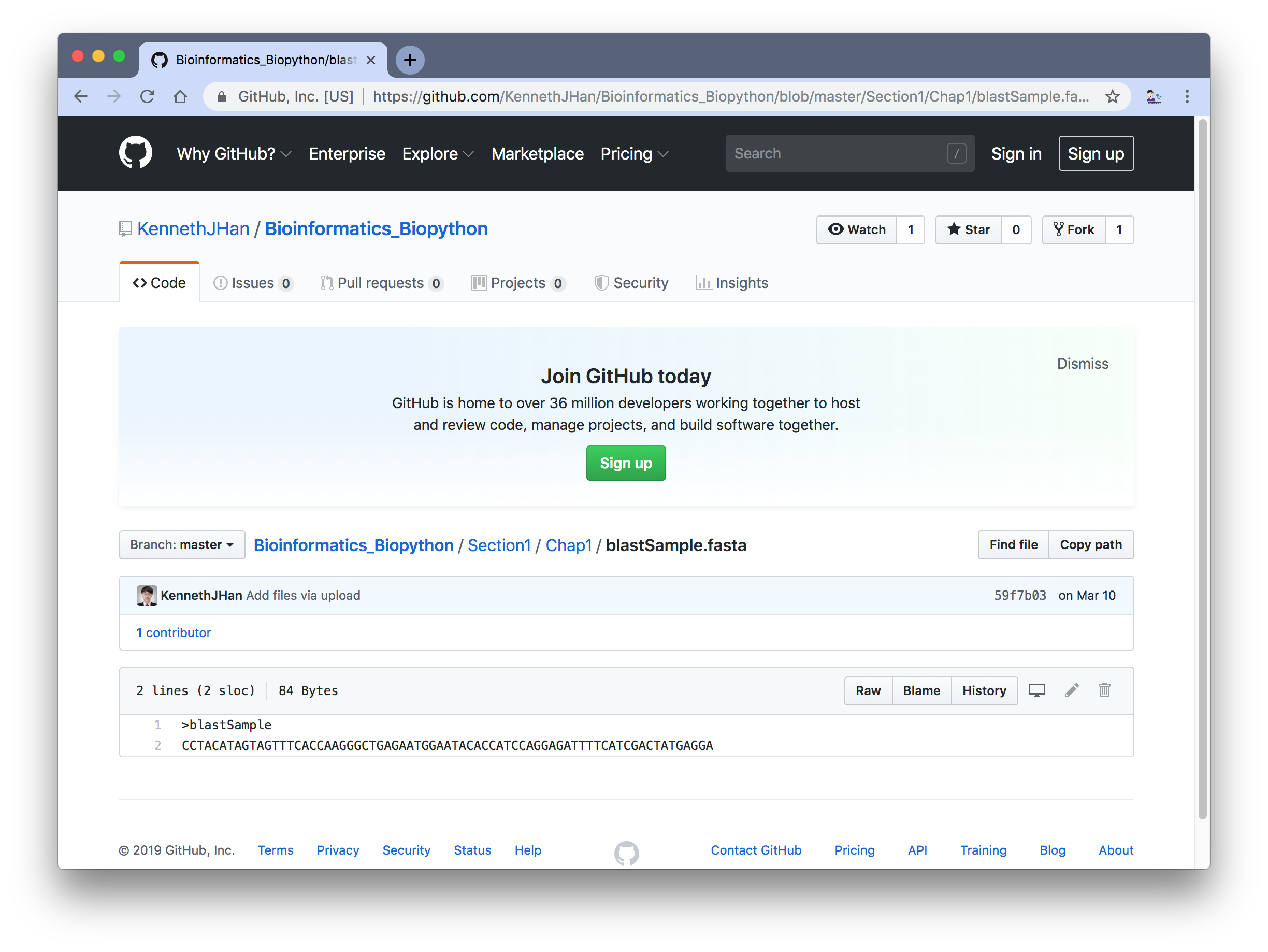
파일의 내용 윗 부분에 Raw 라고 된 버튼을 눌러보겠습니다. 그러면 다음 그림과 같이 파일의 raw한 상태가 나오는데요..
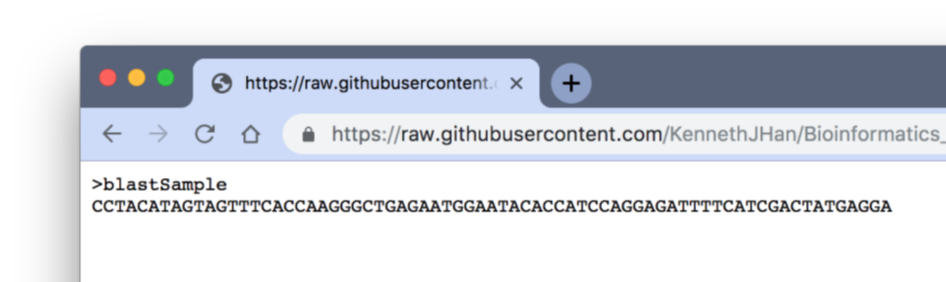
이 상태를 저장해주시면 됩니다.
윈도우라면 Ctrl 과 S 키를 눌러주시고, 맥이면 Cmd 와 S 키를 눌러주세요.
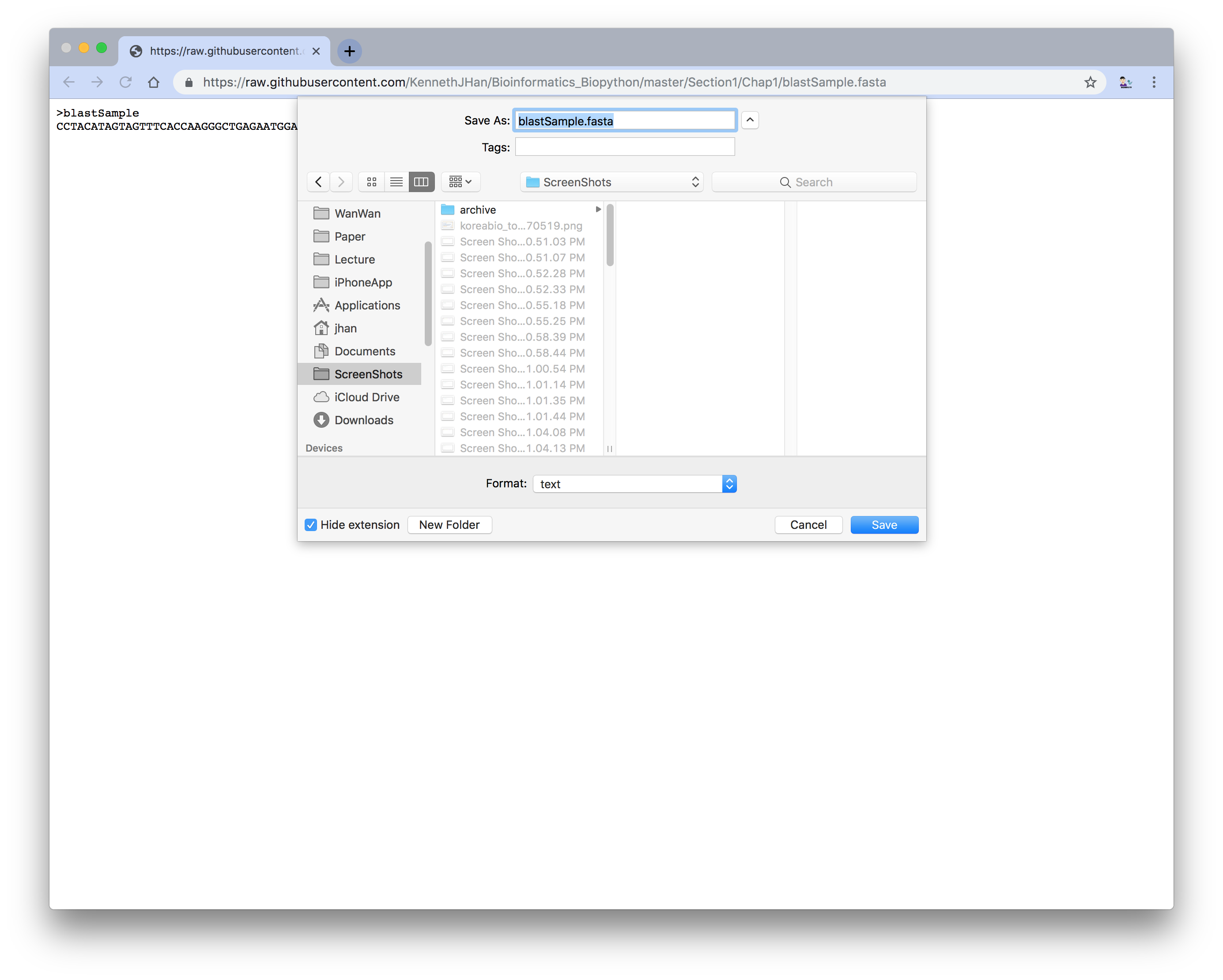
적당한 장소에 저장해 주심 됩니다 ㅎㅎ
2. 한 번에 zip으로 모든 파일 받기
파일을 하나씩 받을 때는 1번에서 보여드린 방법대로 하면 됩니다만,
모든 파일을 받아야 하는 경우라면.. 매번 하나씩 받기에는 힘들겠지요?
그래서 한 번에 zip으로 받는 방법이 있습니다.
다음 페이지에 들어가셔서 초록색 버튼의 Clone or download 버튼을 눌러줍니다.
https://github.com/KennethJHan/Bioinformatics_Biopython

누르면 Download ZIP 버튼이 나오며 이걸 눌러주면 한 번에 모든 파일들을 받으실 수 있습니다.
3. git clone 명령어로 받기
마지막으로 콘솔 환경에서 받는 방법에 대해 말씀드리겠습니다.
우리는 git이란 명령어를 이용하여 github 저장소에 있는 파일들을 그대로 복제 (clone) 할겁니다.
윈도우에서는 기본적으로 git 명령어가 없어서 (윈10에서는 이제 리눅스 명령어를 쓸 수 있다고 합니다..?)
git bash와 같은 프로그램을 설치하셔야 합니다. 다음 주소를 참고해주세요.
리눅스나 맥에서는 기본적으로 git명령어가 설치 되어있습니다.
준비가 되셨다면 콘솔에서 다음과 같이 명령어를 입력 후 엔터를 치시면 됩니다.
$ git clone https://github.com/KennethJHan/Bioinformatics_Biopython.gitgit clone 뒤 주소는 Clone or download를 눌러서 나온 화면에 주소를 복사하시면 되는데,
드래그 하셔서 카피하셔도 되고 , 또는 주소 옆의 문서 아이콘 같이 생긴.. 버튼을 누르시면 자동으로 복사가 됩니다.

오늘은 github에서 자료를 다운로드 받는 방법에 대해 알아봤습니다.
모든 지식이 그렇겠지만,, 알고나면 쉬운데 처음이 어렵습니다 ^^;;
부디 여러분들께 도움되셨음 좋겠네요
그럼 다음 시간에 만나요 ~~~~
'생물정보학 > Tools' 카테고리의 다른 글
| BED 파일을 ucsc genome browser에 추가하는 방법 (0) | 2019.10.06 |
|---|---|
| VCF GT, DP, AD와 genotype 0/0 , 0/1 , 1/1의 의미 (1) | 2019.07.15 |
| [Tool] BLAST 리눅스 로컬 설치 및 실행방법 - BLAST LINUX local install and execute - BLAST 로컬 설치의 장단점 (2) | 2018.12.08 |
| [minimap2] minimap2 설치 및 수행 방법 (0) | 2018.11.18 |
| [Tool] gnomAD 란? gnomAD vcf 다운로드 방법 및 압축 푸는 방법- gnomAD 활용 방법 - gnomad vcf bgz: unknown suffix -- ignored (2) | 2018.11.17 |



댓글