안녕하세요
한주현입니다
오늘은 ORF (Open Reading Frame)와 CDS (Coding Sequence) 의 차이점과 refGene.txt.gz 파일에 대해서 알아보겠습니다.
ORF
ORF (Open Reading Frame)는 시작 코돈 (AUG)에서부터 종결 코돈 (UAA, UAG, UGA) 까지의 서열을 말합니다.
ATG [xxx] TAG 와 같이 시작과 종결 사이에 triplet code, 즉 3배수의 염기들로 구성 되어만 있다면 ORF 라고 부를 수 있습니다.
실제로 DNA 서열에서 ATG (3n) TAG 로 구성된 ORF 서열을 찾아보면 많이 존재합니다.
이러한 ORF를 찾아주는 프로그램들도 존재하지요.
1) ORF Finder
https://www.bioinformatics.org/sms2/orf_find.html
2) ORF Investigator
https://sites.google.com/site/dwivediplanet/ORF-Investigator
3) ORF Predictor
https://bioinformatics.ysu.edu/tools/OrfPredictor.html
4) NCBI - ORFfinder
https://www.ncbi.nlm.nih.gov/orffinder/
CDS
CDS (Coding Sequence)는 이러한 ORF 들에서 실제로 Protein으로 번역되는 서열을 의미합니다.
Eukaryote에서는 exon과 intron이 있는데 CDS는 intron을 제외하고, 시작 코돈부터 종결 코돈까지의 서열을 말합니다.
그림으로 보면 훨씬 이해가 쉬우니 그림으로 다시 한 번 알아보죠.
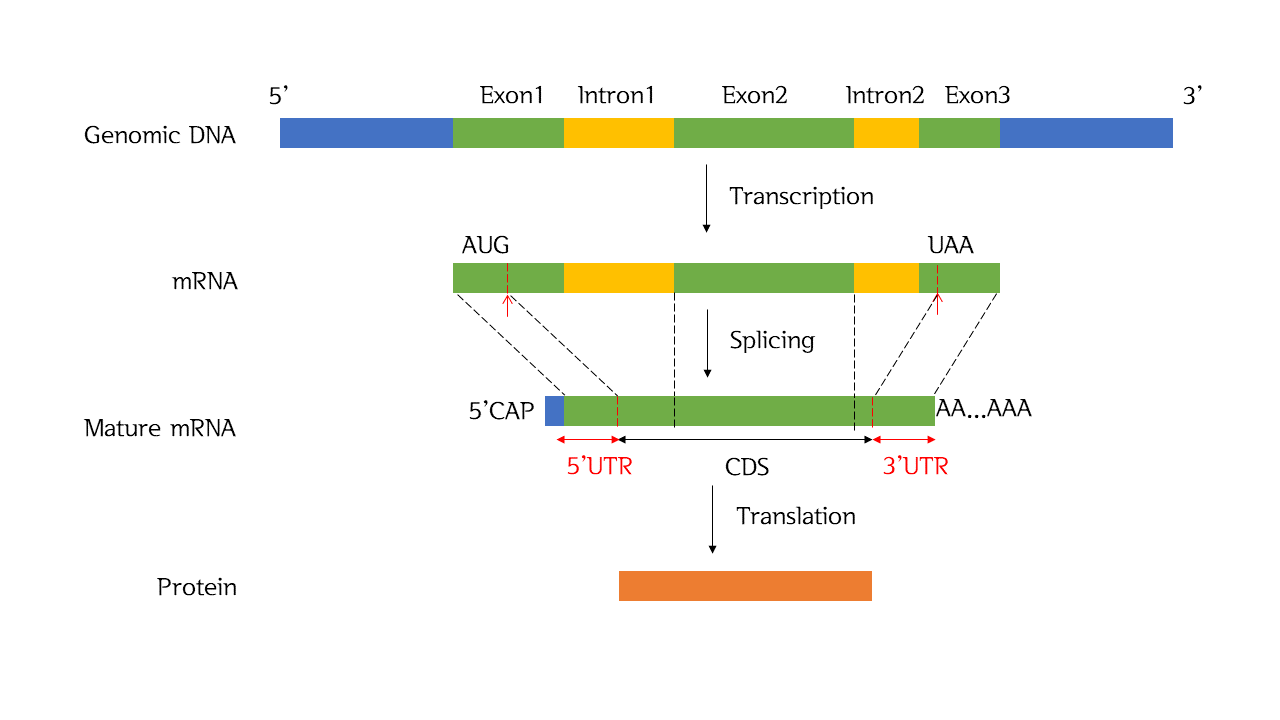
Genomic DNA에서 전사(transcription)가 일어나서 mRNA가 됩니다. 이때 Exon안에서 시작 코돈(AUG)과 종결 코돈(UAA)이 있습니다.
그림에서는 시작코돈이 Exon1, 종결코돈이 Exon3에 들어있는데 꼭 처음과 마지막에 들어있을 필요는 없습니다.
시작 코돈이 Exon1이 아니고 Exon2에 있을수도 있고, 종결 코돈이 Exon3이 아니고 Exon2에 있을수도 있습니다.
Splicing 과정을 거치면 Mature mRNA가 되고 이때 시작 코돈과 종결 코돈 사이를 CDS라고 부릅니다.
이 CDS가 번역(translation)이 되어 단백질이 됩니다.
refGene.txt
UCSC 에서 제공하는 refGene 파일은 reference gene 정보를 제공합니다.
아래 링크로 들어가보면 UCSC에서 제공하는 여러 파일들이 있는데,
http://hgdownload.cse.ucsc.edu/goldenpath/hg38/database/
Index of /goldenpath/hg38/database
hgdownload.cse.ucsc.edu
http://hgdownload.cse.ucsc.edu/goldenpath/hg19/database/
Index of /goldenpath/hg19/database
hgdownload.cse.ucsc.edu
링크에 들어가서 다음 그림과 같이 refGene.txt.gz 파일을 받습니다.

gz 확장자는 gzip이라는 형식으로 압축된 파일입니다.
파일을 살펴볼까요?
윈도우 사용자는 7-zip 과 같은 프로그램으로 압축을 해제해줍니다.
리눅스 커맨드라인이 사용가능하신 분들은 그냥 zless로 보면 됩니다.
이번 글에서는 엑셀로 한 번 열어보겠습니다. 엑셀로 열기를 누르고 텍스트 파일 그 자체를 열어보시면 됩니다.
주의할 점이 두 가지가 있는데,
문서의 열이 탭으로 구분되어있기에 구분자를 탭으로 설정하는 것과
Gene 이름이 있는 컬럼은 반드시 Text 로 설정하셔야 한다는 것입니다.
만약 Text가 아니고 기본 설정인 General로 하면 MARCH1 유전자는 3월1일.. SEPT2 유전자는 9월2일.. 과 같이 엑셀이 친절하게 자동으로 바꿔줍니다.. ㅋㅋ
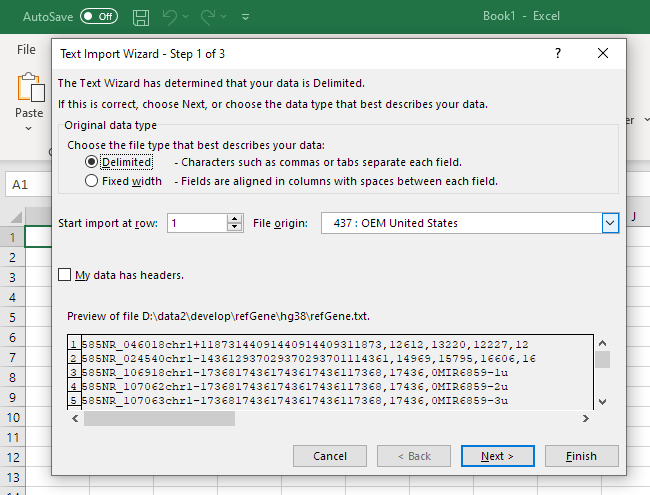



위 스크린샷에서 첫 번째 행은 제가 넣은 행입니다.
refGene의 컬럼에 대한 자세한 설명은 다음 링크를 참고해주세요.
http://genome.ucsc.edu/FAQ/FAQformat#format9
그냥 글을 마무리 하면 아쉬우니 refGene에서 한 행을 그림으로 표시하면서 어떠한 의미인지 알아보겠습니다.
예시로 만들 행은 ZNF595 유전자입니다.

보기가 좀 복잡해 보일 수 있는데, txStart 부터 차근차근 하나씩 살펴보면 그렇게 복잡하지 않습니다.
txStart는 transcript가 시작하는 지점,
txEnd는 transcript가 끝나는 지점입니다.
mRNA가 시작하는 지점을 Genomic DNA에 53284, 88211 로 표시하였습니다.
cdsStart는 coding sequence가 시작하는 지점,
cdsEnd는 coding sequence가 끝나는 지점입니다.
Mature mRNA에서 CDS로 표시한 부분에 53488, 87541로 표시하였습니다.
마지막으로 exonStarts, exonEnds가 있습니다.
이는 각 exon들이 시작하는 지점들과 끝나는 지점들을 쉼표로 구분한 데이터로
mRNA 쪽에 각각 위치를 표시하였습니다.
오늘은 ORF와 CDS 그리고 refGene.txt 파일에 대해 살펴보았습니다.
그럼 다음에 또 만나요~
'생물정보학 > Tools' 카테고리의 다른 글
| [Tool] Expansion Hunter 설치 및 사용방법 (0) | 2022.11.24 |
|---|---|
| [VCF 파일] genozip - VCF 파일의 효율적인 압축 방법을 제시한 툴 (0) | 2020.07.12 |
| [생물정보학 유튜브 강의] 생물정보학 툴 설치 (samtools) 및 bam 파일 보는 방법 (1) | 2020.06.14 |
| BED 파일을 ucsc genome browser에 추가하는 방법 (0) | 2019.10.06 |
| VCF GT, DP, AD와 genotype 0/0 , 0/1 , 1/1의 의미 (1) | 2019.07.15 |




댓글