안녕하세요
한주현입니다
생물정보학 코딩 연습과 파일 형식을 익히기 위한 포스팅 두 번째 시간입니다
오늘은 VCF를 활용한 생물정보학 문제를 포스팅하겠습니다
문항) VCF파일에서 SNP, InDel count하기
VCF (Variant Calling Format) 파일은 텍스트 형식의 파일로 meta-information lines, Header, data lines 로 구성되어있습니다
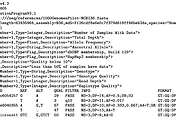
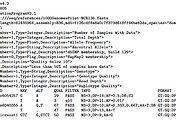
VCF의 Data Line에서 4번째 컬럼과 5번째 컬럼인 REF와 ALT를 보겠습니다.
REF는 Reference base(s)로 hg19, hg38 과 같이 기준이 되는 서열에서 각 해당 position에서의 서열을 의미합니다.
ALT는 Alternate base(s)로 sequencer 에서 읽어낸 base가 REF 와 다를 경우 나타납니다.
예를 들어 위의 VCF 예제 그림에서 chr20 14370 에서 REF는 G, ALT는 A로 쓰여있습니다.
SNP(Single Nucleotide Polymorphism)은 VCF에서 1개의 base의 change로 나타납니다.
예를 들어 위의 VCF 예제 그림에서 chr20 14370 에서 REF는 G, ALT는 A로 쓰여있는 것 처럼 하나의 base가 하나의 base로 바뀐 경우 SNP라고 합니다
Insertion은 VCF파일에서 REF의 길이보다 ALT의 길이가 더 길게 표현합니다.
Deletion은 VCF파일에서 REF의 길이보다 ALT의 길이가 더 짧게 표현합니다.
SNP와 InDel은 아래 표와 같은 예시로 정리 할 수 있습니다.
Type |
REF |
ALT |
SNP |
A |
C |
INS |
T |
TA |
DEL |
ACT |
A |
이번 문항에서는 VCF에서 SNP와 Insertion, Deletion의 개수를 세어 보겠습니다.
문항에 해당하는 VCF는 아래의 VCF를 사용하세요.
https://raw.githubusercontent.com/KennethJHan/Bioinformatics_smalltalk_Python50/master/sampleVCF.vcf
VCF에 대한 자세한 설명은 아래 링크를 참고하시기를 바랍니다.
https://samtools.github.io/hts-specs/VCFv4.2.pdf
정답)
SNP: 14
Insertion: 2
Deletion: 4
문항에 의문사항이 있으시면 댓글로 말씀 부탁드리겠습니다
그럼 다음 문항에서 만나요~
'생물정보학 > 생물정보학_연습문제' 카테고리의 다른 글
| [Sequence] 04. DNA 서열에서 base count 하기 (1) | 2017.04.07 |
|---|---|
| [VCF] 03. VCF 파일에서 Variant 개수 count 하기 (1) | 2017.04.06 |
| [VCF] 01. VCF 파일에서 PASS filter 하기 (3) | 2017.04.03 |
| [공지] 생물정보학 연습문제 카테고리를 시작하며 (2) | 2017.04.03 |

댓글