반응형
안녕하세요 한주현입니다.
오늘은 vcf 파일을 chromosome 별로 position 별로 정렬 sorting 하는 방법에 대해 알아보겠습니다.
| 들어가며 |
가끔 vcf 파일이 다음과 같이 sorting 되어있지 않는 경우가 있는데요,
이렇게 정렬이 되어있지 않으면 어떤 툴들에서는 정렬이 안되었다고 오류가 띡! 뜹니다 ...
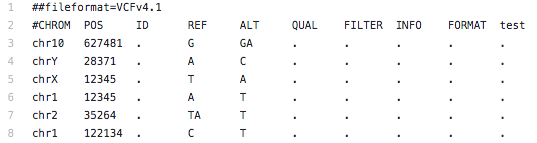
정렬이 안된 vcf 파일을 다음과 같이 정렬하고 싶은거죠.

참고로 vcf 파일은 임의로 생성하였고
편의상 chromosome, position, ref, alt 만 값을 넣었고 나머지는 점(.) 으로 채웠습니다.
| VCF 정렬하기 |
vcf를 정렬하는 방법에는 여러가지가 있습니다만,
프로그램 쓰지 않고 간단히 정렬하는 방법을 찾으시는 분들께,
다음의 리눅스 기본 명령어로 간단히 vcf 파일을 정렬하는 방법이 참~ 유용할 겁니다.
정렬에 사용할 vcf 실습 파일은 다음 github에 있습니다.
https://github.com/KennethJHan/vcf_sort
그러면 out.vcf 는 다음과 같은 형태로 나오게 되지요.

툴을 사용하지 않고 간단히 리눅스 명령어로 vcf 파일을 정렬하는 방법에 대해 알아보았습니다.
여러분들께 도움이 되셨으면 좋겠네요.
그럼 다음에 또 만나요~
반응형
'생물정보학' 카테고리의 다른 글
| [생물정보학 특허] VCF 파일 활용 :: 유전자 서열 변이 정보를 활용한 혈연 관계 판단 방법 :: 특허 등록 후기 (0) | 2022.05.22 |
|---|---|
| [번역서 출간] 생물정보학 알고리듬 3rd edition, 번역 후기 (9) | 2022.03.01 |
| 생물정보학 서버 사양 - 클라우드 vs. 온프레미스 (2) | 2022.01.25 |



댓글