안녕하세요 한주현입니다.
오늘은 바이오파이썬 1.78에서 달라진 Seq 클래스에 대해 알아보겠습니다.
들어가며
2020년 9월에 바이오파이썬의 1.78 버전 업데이트로,
Seq 클래스의 사용법이 조금 바뀌었습니다.
오늘은 1.78 버전에서 Seq 클래스의 달라진점과 도서에서 작동하지 않는 부분을 어떻게 하면 실행할 수 있는지 방법에 대해 알아보겠습니다.
버전업!
앞서 말씀드렸듯이 2020년 9월 바이오파이썬이 1.78로 업데이트 되었습니다.
덕분에 ㅎㅎ.. 도서에서 사용했던 코드가 작동하지 않는 부분이 있습니다. ㅜㅜ
다음은 도서 70페이지의 4.5.2 Bio.SeqUtils로 서열의 무게를 계산하는 코드인데요,
ATGCAGTAG를 DNA 로 볼 경우와 아미노산 서열로 보는 경우에 따라 다르게 무게가 계산이 되는 tricky 하고 심도있는! 예시를 야심차게! ㅎㅎ.. 넣어두었습니다. 서열 겉 보기에는 같지만 DNA와 amino acids 기 때문에 다르다!
from Bio.Seq import Seq
from Bio.Alphabet import IUPAC
from Bio.SeqUtils import molecular_weight
seq1 = Seq("ATGCAGTAG")
seq2 = Seq("ATGCAGTAG", IUPAC.unambiguous_dna)
seq3 = Seq("ATGCAGTAG", IUPAC.protein)
print(molecular_weight(seq1)) # 2453.82 가 출력 됨
print(molecular_weight(seq2)) # 2842.82 가 출력 됨
print(molecular_weight(seq3)) # 707.75 가 출력 됨
그런데 독자분들께서 만약 바이오파이썬 1.78 버전을 설치하여 예시를 따라하신다면 다음과 같은 오류를 만나게 되실겁니다.
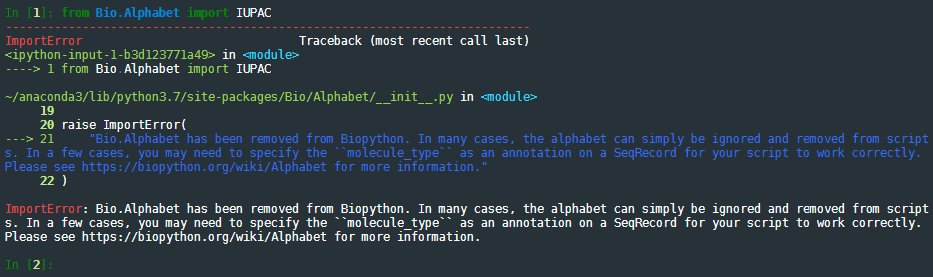
오류는 다음 문장에서 발생하는데요,
from Bio.Alphabet import IUPACImportError 가 발생하면서 실행되지 않습니다.
근데 이거 굳이 왜 바꿨나요?
위의 오류를 보면 information 이라고 하면서 다음과 같은 링크를 하나 줍니다.
History and replacement of Bio.Alphabet · Biopython
History and replacement of Bio.Alphabet Introduction This page is intended to help people updating existing code using Biopython to cope with the removal of the Bio.Alphabet module in Biopython 1.78 (September 2020). The objects from Bio.Alphabet had two m
biopython.org
들어가보시면 Seq 객체에서 더 이상 type을 지정하지 않는 방향으로 바꾸었다고 했습니다.
실제로 IUPAC DNA 로 지정하면 A, C, G, T 의 문자만 허용하여 개발상 제약이 있던 문제가 있었습니다.
제가 개발할 때 겪었던 문제를 예시로 들어보면, 서열에서 gap 이 있는 경우 -, *, = 등 과 같은 다양한 문자로 표현하기도 하는데 이때마다 Seq을 초기화 할 때 동적으로 넣는것이 불가능 한 경우를 맞딱뜨린 경우가 있었습니다. ㅋㅋ.
그럼 어떻게 하면 도서의 코드를 활용할 수 있을까요?
먼저 BioAlphabet 에서 IUPAC 을 사용하는 부분을 살펴보겠습니다.
58페이지의 4.3.2 Alphabet 모듈은 바이오파이썬 버전 1.78 에서는 사라졌기에 참고로만 봐주세요.
[스크립트 4-2] Alphabet 모듈의 코드는 다음과 같이 수정하시면 됩니다.
from Bio.Seq import Seq
# from Bio.Alphabet import IUPAC # 이 라인을 삭제합니다.
# tatabox_seq 라인을 다음 문장과 같이 IUPAC.unambiguous_dna 부분을 삭제합니다.
# tatabox_seq = Seq("tataaaggcAATATGCAGTAG", IUPAC.unambiguous_dna) # 원본
tatabox_seq = Seq("tataaaggcAATATGCAGTAG") # 수정
print(tatabox_seq) # tataaaggcAATATGCAGTAG 가 출력된다.
print(type(tatabox_seq)) # <class 'Bio.Seq.Seq'>
70페이지의 4.5.2 Bio.SeqUtils로 서열의 무게 계산하기 에서 [스크립트 4-16] Bio.SeqUtils로 서열의 무게 계산하기 는 다음과 같이 수정하시면 됩니다.
from Bio.Seq import Seq
# from Bio.Alphabet import IUPAC # 삭제
from Bio.SeqUtils import molecular_weight
seq1 = Seq("ATGCAGTAG")
# seq2 = Seq("ATGCAGTAG", IUPAC.unambiguous_dna) # 삭제
# seq3 = Seq("ATGCAGTAG", IUPAC.protein) # 삭제
print(molecular_weight(seq1)) # 기본적으로 sequence type은 DNA 입니다.
# print(molecular_weight(seq2)) # 삭제
# print(molecular_weight(seq3)) # 삭제
print(molecular_weitght(seq1, seq_type="DNA") # seq_type="DNA"로 DNA를 명시적으로 설정하였습니다.
print(molecular_weitght(seq1, seq_type="protein") # seq_type="protein" 으로 설정하였습니다.
# 위 스크립트를 실행하면
# 2842.82
# 2842.82
# 707.75
# 가 출력됩니다.
바이오파이썬에서 Alphabet 모듈을 제거하였기에 코드와 같이 이 부분을 수정하셔서 보시거나
여러분들께서 바이오파이썬 1.77 이하 버전을 설치하셔서 사용하시면 됩니다 ㅎㅎ;;
참고로 pip 에서 라이브러리 특정 버전으로 설치하는 방법을 말씀드리겠습니다.
다음과 같이 커맨드라인에서 pip install biopython==1.77 을 입력해줍니다.
pip install biopython==1.77
그러면 바이오파이썬 1.77이 설치되고..
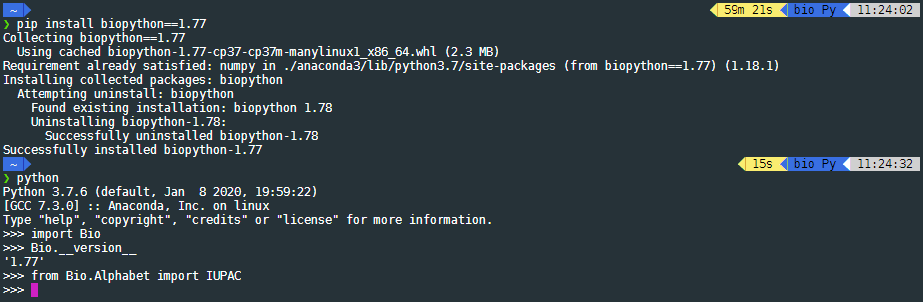
도서와 같이 Bio.Alphabet 의 IUPAC을 문제없이 사용할 수 있습니다!
궁금하신점 댓글로 남겨주시면 답변드리겠습니다~
p.s. 댓글로 제보주신 YHT 님 감사합니다!

그럼 모두들 화이팅 하시고 다음에 또 만나요~
'생물정보학 > Biopython (바이오파이썬)' 카테고리의 다른 글
| [생물정보학 유튜브 강의] 코로나 바이러스 covid 19 서열로 웹로고 그려보기 (0) | 2020.06.21 |
|---|---|
| [바이오파이썬] 바이오파이썬 설치와 jupyter notebook에서 실행하는 방법 (5) | 2020.06.07 |
| [생물정보학 파이썬] 코로나 바이러스-19 (COVID-19) 서열 분석, WebLogo, Multiple Sequence Alignment, 계통수 (phylogenetic tree) 그리기 (10) | 2020.03.01 |
| [도서] 바이오파이썬으로 만나는 생물정보학 (26) | 2019.03.12 |
| [바이오파이썬] 5.1.1 SeqIO 모듈로 서열 파일 읽기 - FASTA (12) | 2018.12.24 |




댓글